シークエンス解析における酸化ストレスによるアーチファクトについて
DNA damage is a pervasive cause of sequencing errors, directly confounding variant identification
最近Scienceに、酸化ストレスによる生じる変異コールのアーチファクトについての論文が出ました。 シークエンス解析において、検体によっては、酸化ダメージによるG>T(C>A)の変異(oxoG)が頻発する場合があることが知られているのだが、 これについてのいくつかの解析を加えた論文です。
インターネットの反応を見ると、いくつかの批判が出ているようです。
批判の主な主張としては、
- 2013年のNARの論文に出ていた内容を再現しただけ。
- 既にシークエンス解析のコミュニティーではこちらの現象は知られており、この現象によるアーチファクトをフィルターすることも(後で説明します)、多くの大規模がんゲノム解析で既に普通に行われている。
というものです。そして、自分もその批判に基本的に賛成です。 さらに言うと、一応この論文では、TCGAのシークエンスデータを解析して、「78%のがんでG>Tの偽陽性は50%以上」という見積もりを出していますが、その解析でも以下のようないくつかの重大な問題があります。
- もともとG>Tの変異(タバコによるシグナチャー)が多く生じているLung Adenocarcinomaを使って解析しており、本物の変異とoxoGのアーチファクトの違いがわからない。oxoGのアーチファクトがあり、かつ本物の変異中にG>Tの変異が少ないもの(Thyroid Cancerなど)で検証するべき。
- 通常するべきフィルタリング(panel of normal)がされていない。p-valueの閾値が緩すぎる(panel of normalを使わず、VarScan2でp-valueを0.05に設定すると、偽陽性率がかなりひどいことになります)。
- 論文でhigh-confidence variantとの記載があるがその定義がどこにも書いていない(stringent filterを突破したものとしか書いていない)。
以上のことを鑑みると、故意にartifactが際立つような、極めて緩い基準での変異コール基準を設定して、「oxoGの変異はたくさんあります!」という主張をしているように思えます。
とりあえず、Scienceにも出ているということで、今後oxoGのアーチファクト関連のことを聞かれることもあると思うので、以下のことを主張しておきたいと思います。
- TCGAのデータに、oxoGによるアーチファクトが大量に生じている検体は確かに生じているが、この論文の主張(78%のがんでG>Tの偽陽性は50%以上)というのは全く嘘。
- oxoGのアーチファクトで著しい問題のある検体はせいぜい2~5%くらいだと思う。
- その問題のある検体についても、そのうちのほとんどは適切なoxoGフィルターを施すことで、ほぼ問題がなくなる。
TCGAのLUAD(Lung Adenocarcinoma)を利用したoxoGのアーチファクトの状況の調査
実際にTCGAのLung AdenocarcinomaでGenomon2により変異コールをして、oxoGのアーチファクトの状況を調査して見た。
まずは、いつも通り変異コールをして、手前味噌の方法によりmutation signature解析をした結果は以下のようになった。 mutation signatureの表現は、下段が変異周辺の塩基の組成(例えばAPOBECでは前の塩基はTである確率がとても高い、といったように)を表しており、真ん中の塩基の上段は、変化塩基のパターンを表している(APOBECでは、C>A, C>Gがほとんど、TobaccoではC>Aがまあまあ多いが、T>A, T>Cなどの変化も少しあり)。 最後に、+, -の2本の棒は、transcribed strand, untranscribed strandの比率を表している。
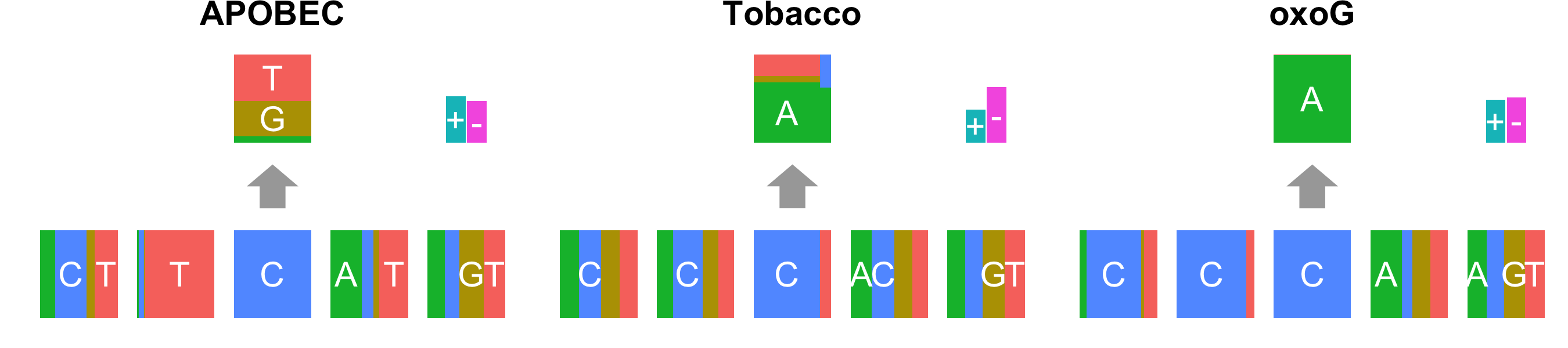
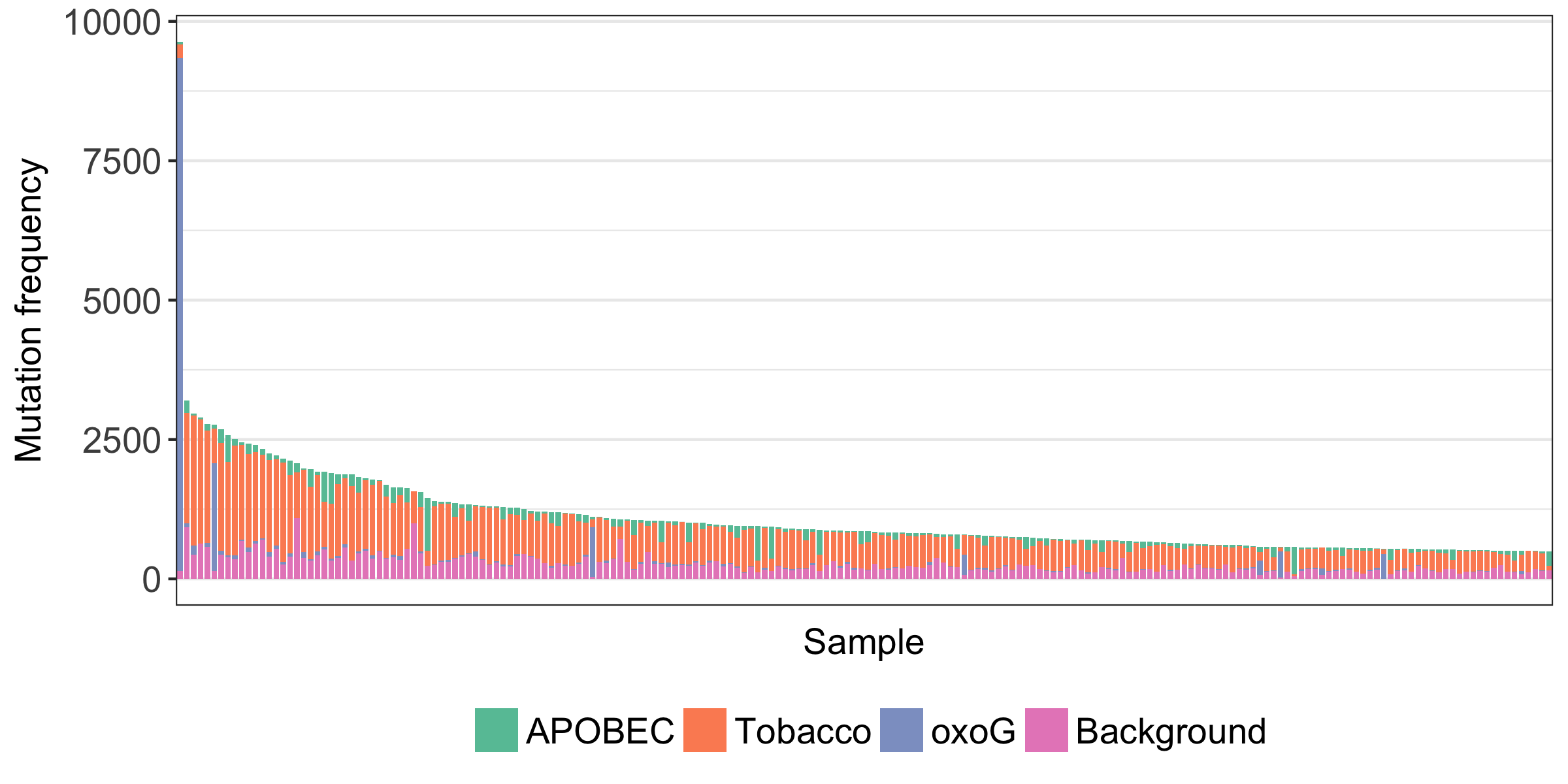
やはり、oxoGのパターンとしては、CpCpN>CpApNと言われているが (実際には前の塩基はCとは限らないと前述のScienceの論文でも指摘されており、それは自分たちの他の解析からの経験でも本当だと思う)、 その通りのmutation signatureが検出された。検体によっては、数千、数万の変異のうち、ほとんどがoxoGのアーチファクトに分類されるものもあった。
では、oxoGのアーチファクトをフィルターするとどうなるか?oxoGのアーチファクトの変異の特徴として、 C>Aの場合はread2, G>Tの場合はread1に集中することが知られている(前述のNARの論文)。 この現象を利用して、変異がG>Tの変異がread1に集中している場合、C>Aの変異がread2に集中している場合はフィルターを施し、 mutation signature解析をすると以下のようになった。
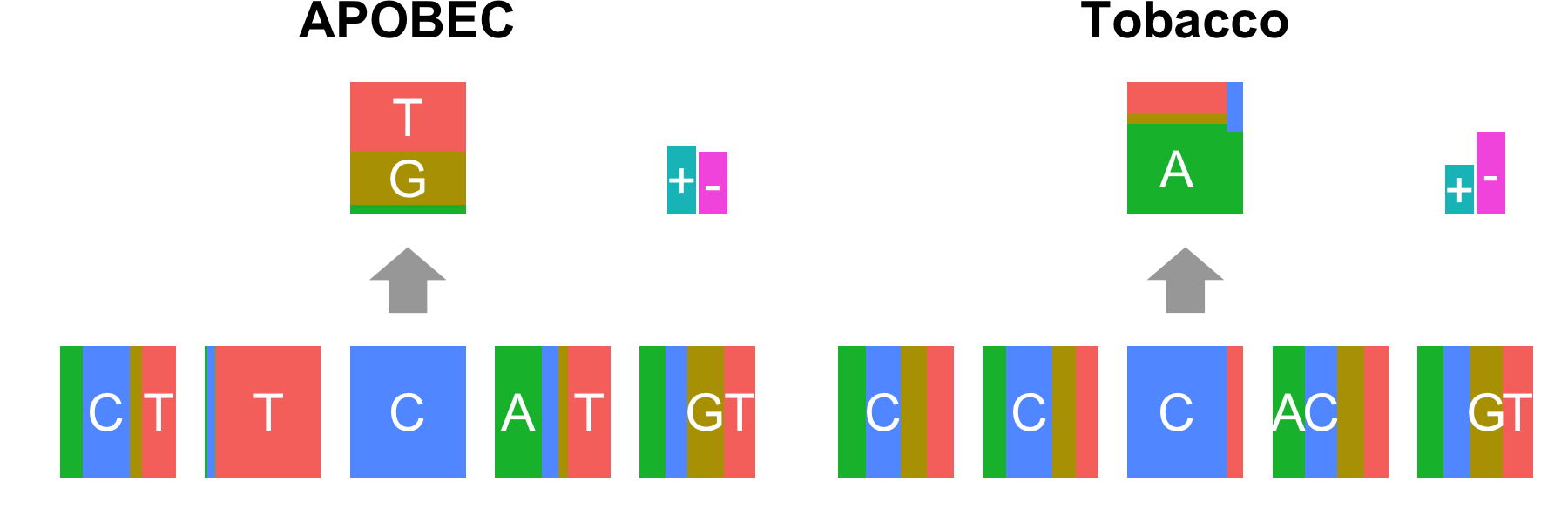
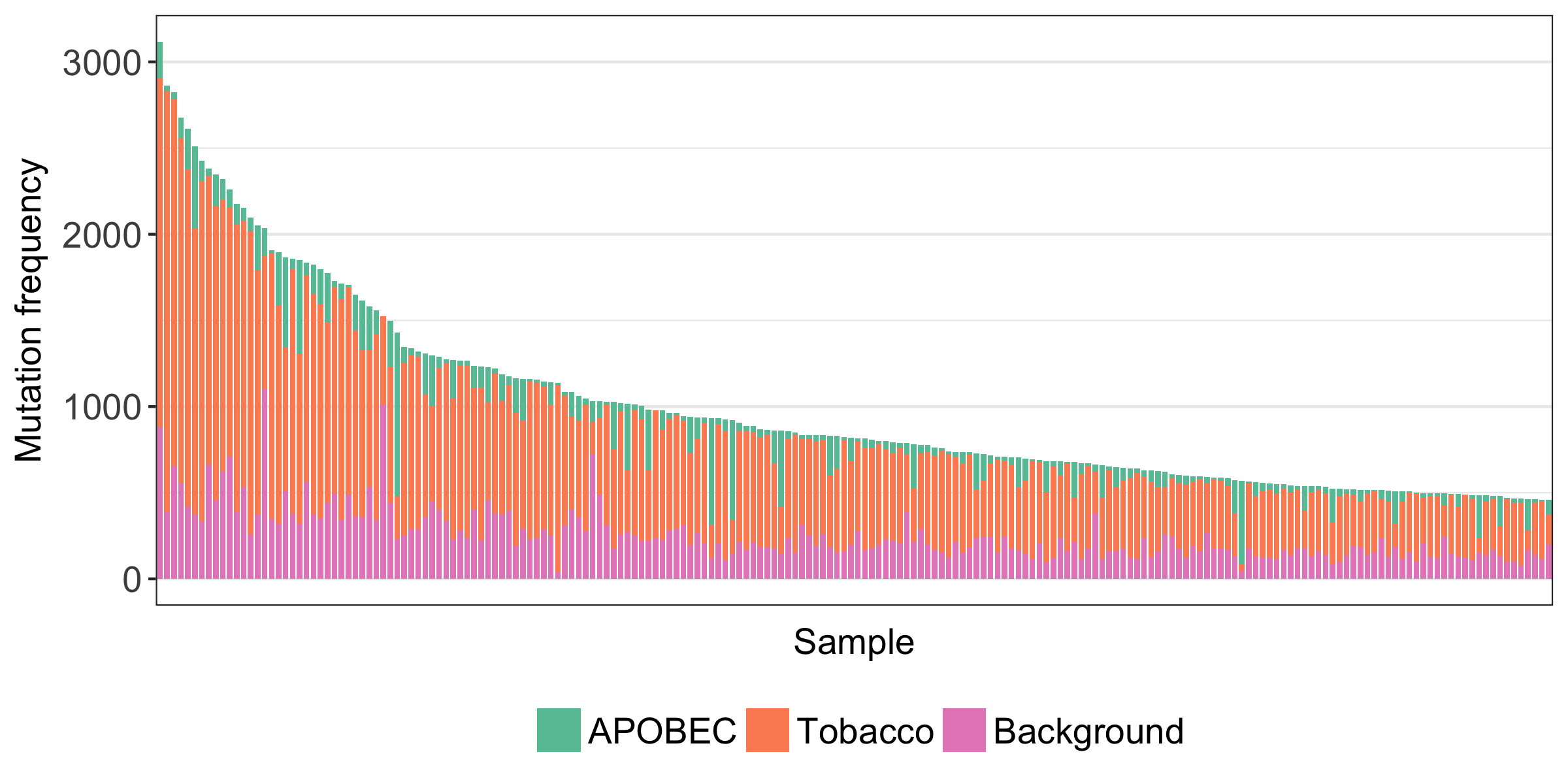
こちらで、oxoGのmutation signatureは不検出となり、ほとんど消えていることがわかる。
それぞれの検体で、oxoGのフィルタリングにより、どのくらいの割合の変異が消えたかを示したヒストグラムを見ると、
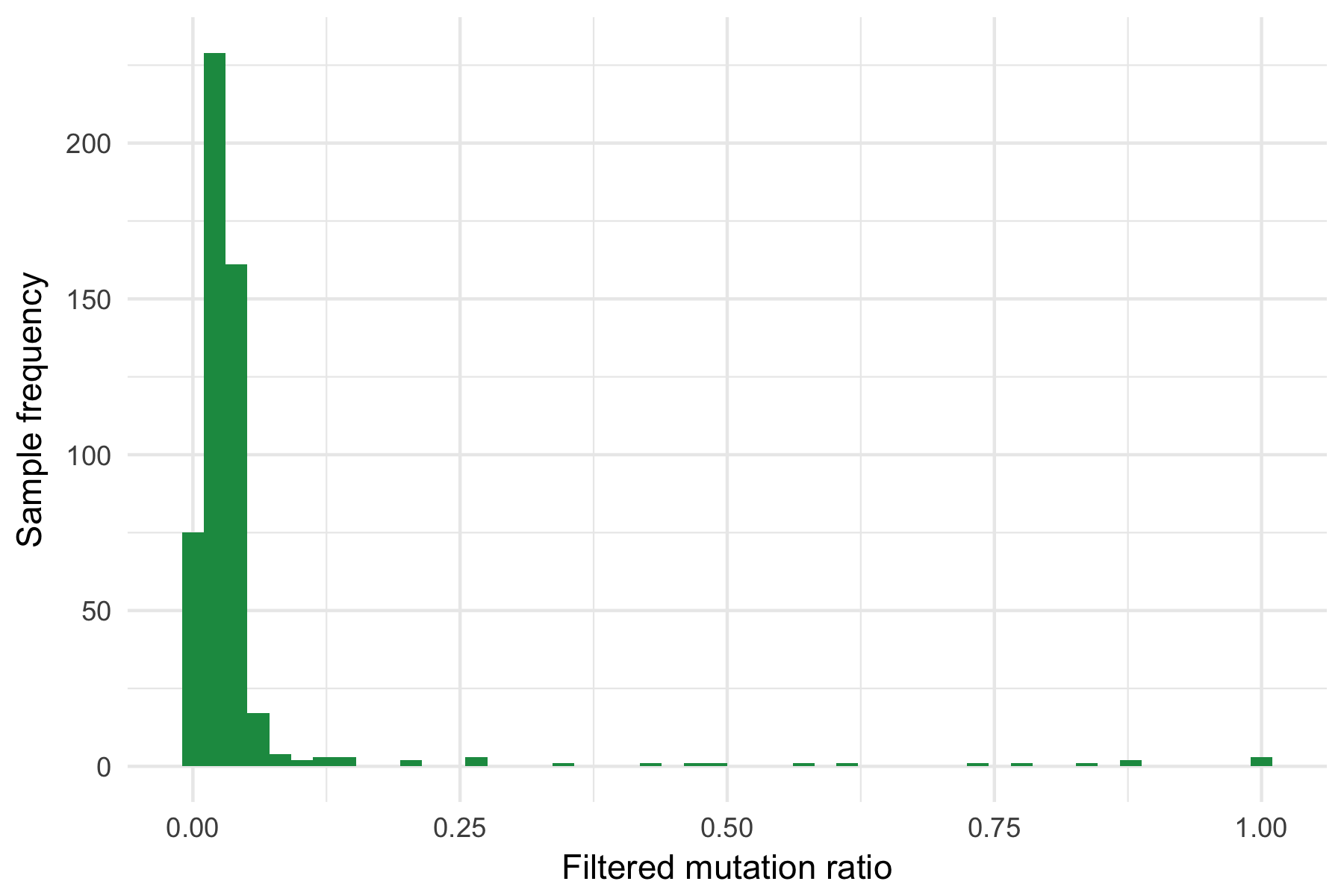 ほとんどの検体では、フィルターされた変異の割合が5%以下にとどまっている
(フィルター率が100%に近いような検体は、oxoGのアーチファクトがものすごい検体です)。
したがって、首尾よくoxoGのアーチファクトを除去しつつ、十分な変異を残し、解析することは全く可能である。
ほとんどの検体では、フィルターされた変異の割合が5%以下にとどまっている
(フィルター率が100%に近いような検体は、oxoGのアーチファクトがものすごい検体です)。
したがって、首尾よくoxoGのアーチファクトを除去しつつ、十分な変異を残し、解析することは全く可能である。
ちなみに、もちろん、こちらの方法でどのくらいの真の変異をフィルタリングしてしまうのか?ということが問題になり (oxoGのアーチファクトの偽陽性ということになるか?)となるが、やはり少し消してしまっているようだ。 とは言っても、COSMICに多く登録のあるようながん遺伝子は基本的には残った。 ただ、唯一、KRASの変異が5個ほど少し消えてしまったという問題点が生じた。 やはり単純にoxoGのフィルターをシステマティックに施すだけではなく、hotspot変異などについては、個別の処理が必要かもしれない。 また、我々は現在、oxoGのフィルタリングの特異性を改善することも取り組んでいるが、 これについてはまた別の機会に詳しく紹介させていただければと思う。
* 2017 4/5, 少し表現がわかりやすくなるように少し追記.
